微流控扩散测量仪—Fluidity One-M
蛋白质分析系统
微流控扩散测量仪Fluidity One-M 是一款用于综合分析蛋白质相互作用以及蛋白质性质的仪器,它采用微流控扩散测量技术(Microfluidic Diffusional Sizing, MDS),可在溶液中一次性测量分子大小、结合亲和力(KD)、浓度和化学计量比。
微流控扩散测量仪产品特点:
-
多参数测量:Fluidity One-M 可以同时测量蛋白质的亲和力、大小、浓度和化学计量比,为研究者提供全面的蛋白质相互作用数据。
-
广泛的样品兼容性:可分析不同缓冲系统中的样品,包括细胞裂解液、血清和唾液等多种生理相关条件下的样本。
-
快速高效:仅需25分钟即可测定24个数据点的KD值,样品消耗量少,每个数据点仅需4 µL,总共50-80 µL即可完成一次KD测定。
-
微流体扩散测量(MDS):利用荧光标记的蛋白质在微流体扩散室中的扩散行为来测定分子的大小。当标记的蛋白与结合伙伴相互作用时,测定到的复合物大小会随之变化,从而进行相互作用分析。
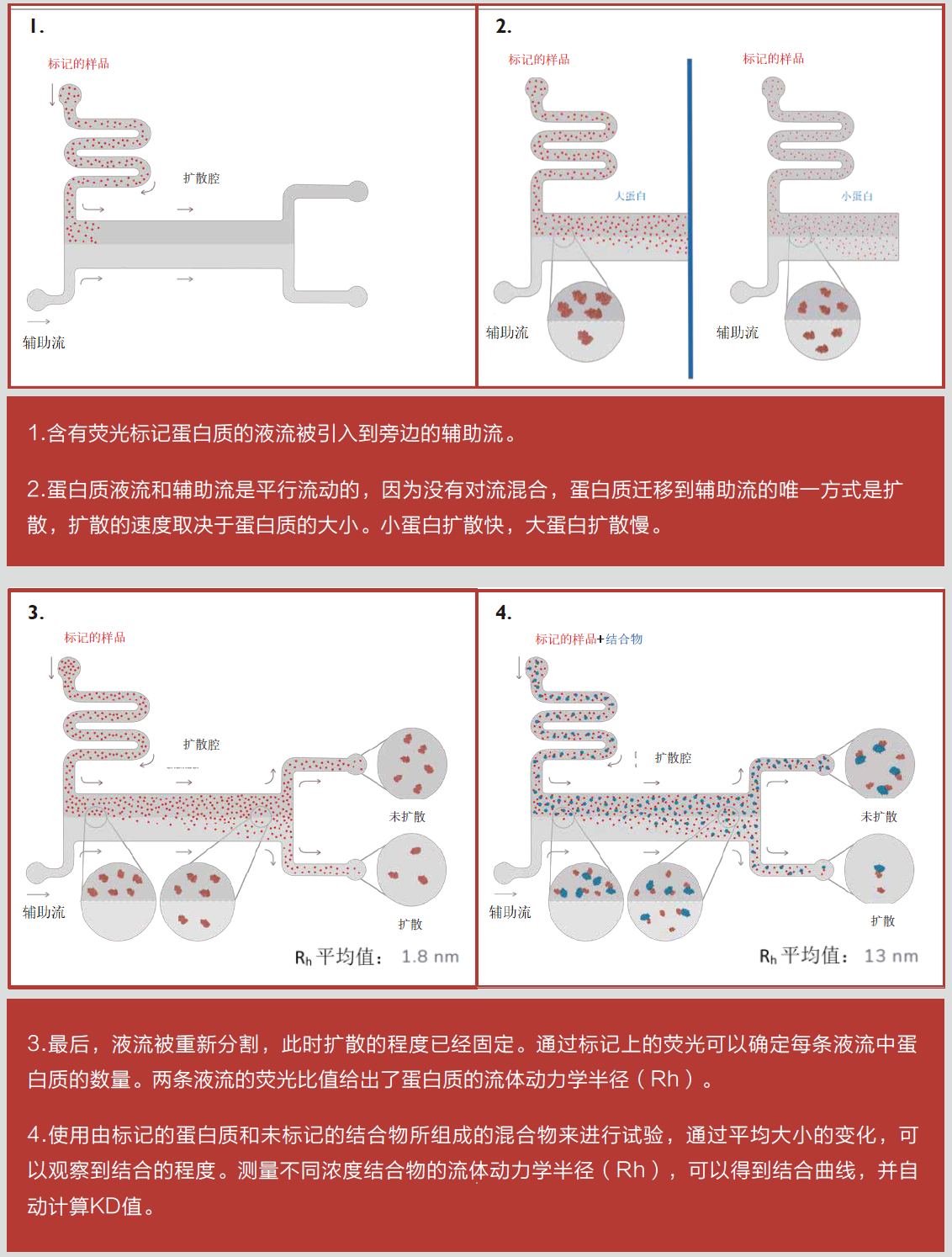
应用领域:
-
基础研究与药物开发:用于研究复杂靶标(如膜蛋白、多蛋白复合物或本质无序蛋白质)的相互作用,甚至可以直接分析粗细胞裂解液、未稀释血清或血浆中的样本。
-
药物筛选与优化:可以在无需纯化的情况下,验证和表征不同靶点的相互作用,并深入了解药物作用机制。
-
过程开发与质量控制:在药物开发过程中,帮助跟踪蛋白质在优化、纯化和制剂开发中的活性变化,并监测蛋白质的粘度和聚集状态。
-
转化和临床研究:帮助测量血清中抗体的浓度和亲和力,并进行药代动力学和血清稳定性研究。
技术优势:
- 无标记直接测量:无需预先校准或生物物理学知识,即可直接测量蛋白质的大小变化,提供定量且易于解释的数据。
- 高灵敏度与特异性:利用荧光检测技术,可在天然环境(如血清和细胞裂解液)中对蛋白质相互作用进行高特异性分析。
- 无维护设计:仪器采用流体无损设计,操作简单,且触屏控制方便,无需频繁维护。
- 云端分析:Fluidity Insight 提供云端实验优化、先进算法分析和跨实验数据集的分析与可视化,帮助研究者更好地理解数据。
技术规格:
- 测量范围:分子水动力半径(Rh)为1 – 20 nm,分子量范围为1.4 kDa – 14 MDa。
- 灵敏度:对于标记的HSA在PBS中的浓度范围,Alexa Fluor 488为1 nM – 3 μM,Alexa Fluor 647为100 pM – 3 μM。
- 兼容性:兼容多种缓冲液和生物缓冲系统,包括TRIS、HEPES、PBS、NaCl、KCl、TWEEN、DMSO和DMF等。
应用案例:
Fluidity One-M 让蛋白质相互作用的分析变得简单高效,为科研工作者提供了一款可靠、便捷且灵活的实验工具,可用于广泛的生物学和药物开发领域。
1. 蛋白质相互作用的结合亲和力(KD)以及流体动力学半径(Rh)
Fluidity One-W利用微流控扩散测量(MDS),可以计算出蛋白质相互作用的结合亲和力(KD)以及流体动力学半径(Rh)。
在该图中,丝氨酸蛋白酶凝血酶与两个预先标记的抗凝血酶配体HD22和TBA进行了滴定。每个配体与凝血酶的不同表位相结合;这些相互作用先前已经被很好地描述过了。每个配体-蛋白质复合物的结合亲和力,以及复合物和配体的流体动力学半径,都符合之前研究报告得到的数值。

2. 天然生物溶液中的蛋白质相互作用
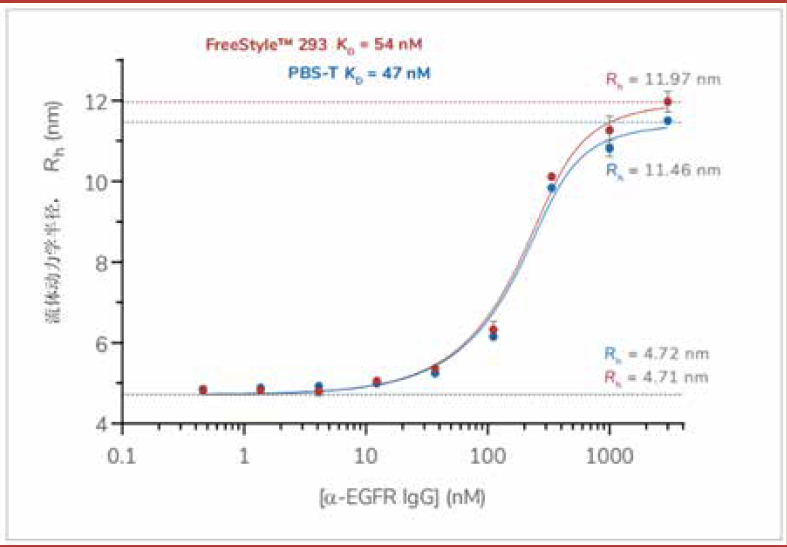
Fluidity One-W可以研究蛋白质在天然生物溶液中的相互作用,节省纯化的时间,也可以在天然条件下研究更困难的相互作用。该图比较了在0.05% 吐温 20 (PBS-T)磷酸盐缓冲液和FreeStyle™293培养基中,A蛋白 (SpA)与免疫球蛋白G (IgG)抗体的相互作用。结果表明,在两种溶液中的结合亲和力和流体动力学半径均无显著差异。
3. 复合物化学计量学的计算
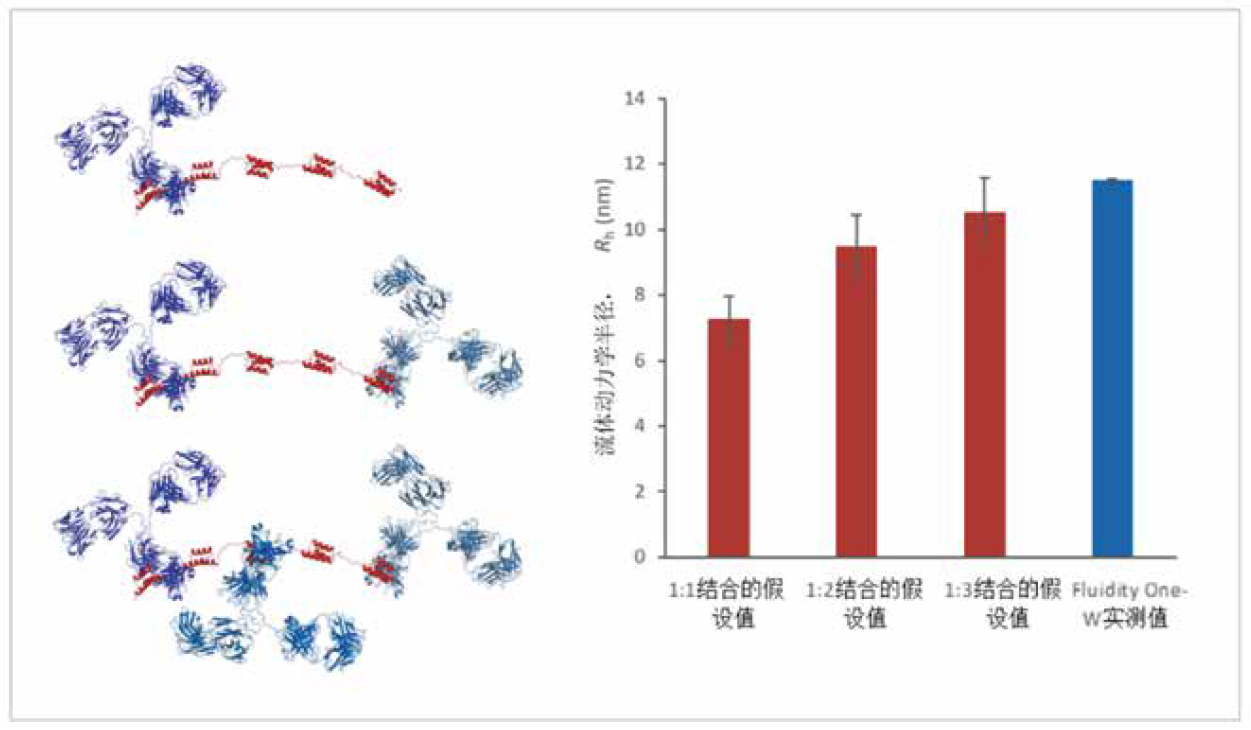
蛋白质复合物化学计量学的研究是充分理解细胞通路的关键;从基础的生物学过程,到疾病病理和药物的作用。在计算结合亲和力(KD)的过程中,Fluidity One-W测量了未结合的标记物和复合物的流体动力学半径(Rh)。流体动力学半径可以用来推断蛋白质复合物的化学计量学和溶液中整体的结合比例与结构。下图的模型模拟了以1:1、1:2和1:3的比例结合的SpA-IgG复合物。红色带状物代表SpA,蓝色带状物代表IgG。利用这些模型计算了三种比例结合物的Rh假设值(红色条形)。Fluidity One-W在100nM浓度下进行3次检测,将测得的Rh平均值(蓝色条形)与这些假设值做比较。红色条形上添加了Rh预测值的10%作为误差范围,以表示模型中的最小不确定性。Fluidity One-W通过微流控扩散测量的Rh值与1:3结合比例的SpA-IgG复合物的Rh假设值相符。对于折叠或延伸的蛋白质,Rh和分子量有各自的换算公式。研究化学计量学的同时,也可以分析蛋白质的结构和分子量。
技术优势:

发表的文献举例
2024
O'Mahoney, Cara et al. "Microfluidic Diffusional Sizing (MDS) Measurements of Secretory Neutralizing Antibody Affinity Against SARS-CoV-2" Annals of Biomedical Engineering 52 (2024): 1653-1664
Read the article
2024
Kelly, Colleen M., Jody L. Martin, and Michael J. Previs. "Myosin folding boosts solubility in cardiac muscle sarcomeres." JCI insight 9.8 (2024).
Read the article
2024
Priddey, Ashley et al. "Microfluidic antibody profiling after repeated SARS-CoV-2 vaccination links antibody affinity and concentration to impaired immunity and variant escape in patients on anti-CD20 therapy" Frontiers in Immunology 14 (2024): 12961...
Read the article
2024
Heuser, Alisa, et al. "Challenges for the Discovery of Non‐Covalent WRN Helicase Inhibitors." ChemMedChem 19.8 (2024): e202300613.
Read the article
2023
Yam-Puc Juan C. et al. "Age-associated B cells predict impaired humoral immunity after COVID-19 vaccination in patients receiving immune checkpoint blockade" Nature Communications 14 (2023):3292
Read the article
2023
Sternke-Hoffmann, Rebecca et al. "Widespread amyloidogenicity potential of multiple myeloma patient-derived immunoglobulin light chains" BMC Biology 21 (2023): 21
Read the article
2023
Shaw, Daniel J. et al. "Modulation of IL-17 backbone dynamics reduces receptor affinity and reveals a new inhibitory mechanism" Chemical Science 14 (2023): 7524-7536
Read the article
2023
Schneider, Matthias M., et al. "Microfluidic antibody affinity profiling of alloantibody-HLA interactions in human serum." Biosensors and Bioelectronics 228 (2023): 115196.
Read the article
2023
Kumar, Suresh et al. "Structural analyses of β2-glycoprotein I: is there a circular conformation?" Journal of Thrombosis and Haemostasis 21 (2023): 3511-3521
Read the article
北京佰司特科技有限责任公司 (https://www.best-sciences.com/)
类器官串联芯片培养仪—HUMIMIC;灌流式细胞组织类器官代谢分析仪—IMOLA;光片显微镜—LSM-200;
蛋白稳定性分析仪—PSA-16;单分子质量光度计—TwoMP;超高速视频级原子力显微镜—HS-AFM;微流控扩散测量仪—Fluidity One-M;
电荷光度计—illumionONE;全自动半导体式细胞计数仪—SOL COUNT;农药残留定量检测仪—BST-100;台式原子力显微镜—ACST-AFM;微纳加工点印仪—NLP2000DPN5000;






